python 对陆地数据进行掩膜的两种方法
1、使用python自带的库进行掩膜2、使用cdo+geopandas+Salem进行掩膜
前提杂谈
一般我们分析海洋或者气象相关数据时,经常会绘制散点图分析两重变量之间的相关性。近期在分析数据时,发现有个容易出问题的情况:
-
如果我们只需要分析海洋上的某一变量的变化时,最好先将陆地数据进行掩膜,防止因为地形等原因导致陆地上的数据存在误差,影响我们的分析结果。
基于上述需求,在这里介绍两种掩膜的方法,这里选择将陆地进行掩膜,掩膜海洋的办法类似。 -
一种是在任何windows系统上的python自带的库进行掩膜,另一种需要通过geopandas+Salem库进行掩膜。其中,后者往往库在Windows上难以安装,一般建议在linux系统进行处理。
方法
- 1、使用python自带的库
global_land_mask进行掩膜 (Windows/Linux都适用) - 2、使用
geopandas+Salem进行掩膜 (建议在Linux系统上使用)
方法1:使用global_land_mask进行掩膜
这里主要通过global_land_mask中的globe.is_ocean()判断数据点的所在的经纬度坐标是否在海洋。如果是陆地,就进行掩膜,反之就不处理。下面封装一个掩膜的函数,方便后续调用处理。
所需要的库主要如下所示:
from global_land_mask import globe
import cmaps
import numpy as np
import xarray as xr
import matplotlib.pyplot as plt
import cartopy.crs as ccrs
from cartopy.mpl.ticker import LongitudeFormatter, LatitudeFormatter
封装掩膜的函数代码如下,其中,对于不同文件的经度名称也进行了考虑设置。
def mask_land(ds, label='land', lonname='lon'):
if lonname == 'lon':
lat = ds.lat.data
lon = ds.lon.data
if np.any(lon > 180):
lon = lon - 180
lons, lats = np.meshgrid(lon, lat)
mask = globe.is_ocean(lats, lons)
temp = []
temp = mask[:, 0:(len(lon) // 2)].copy()
mask[:, 0:(len(lon) // 2)] = mask[:, (len(lon) // 2):]
mask[:, (len(lon) // 2):] = temp
else:
lons, lats = np.meshgrid(lon, lat)# Make a grid
mask = globe.is_ocean(lats, lons)# Get whether the points are on ocean.
ds.coords['mask'] = (('lat', 'lon'), mask)
elif lonname == 'longitude':
lat = ds.latitude.data
lon = ds.longitude.data
if np.any(lon > 180):
lon = lon - 180
lons, lats = np.meshgrid(lon, lat)
mask = globe.is_ocean(lats, lons)
temp = []
temp = mask[:, 0:(len(lon) // 2)].copy()
mask[:, 0:(len(lon) // 2)] = mask[:, (len(lon) // 2):]
mask[:, (len(lon) // 2):] = temp
else:
lons, lats = np.meshgrid(lon, lat)
mask = globe.is_ocean(lats, lons)
lons, lats = np.meshgrid(lon, lat)
mask = globe.is_ocean(lats, lons)
ds.coords['mask'] = (('latitude', 'longitude'), mask)
if label == 'land':
ds = ds.where(ds.mask == True)
elif label == 'ocean':
ds = ds.where(ds.mask == False)
return ds
这个函数调用起来非常简单,只需要输入你的数据,选择掩膜的区域是海洋和陆地,指定经度名称即可。
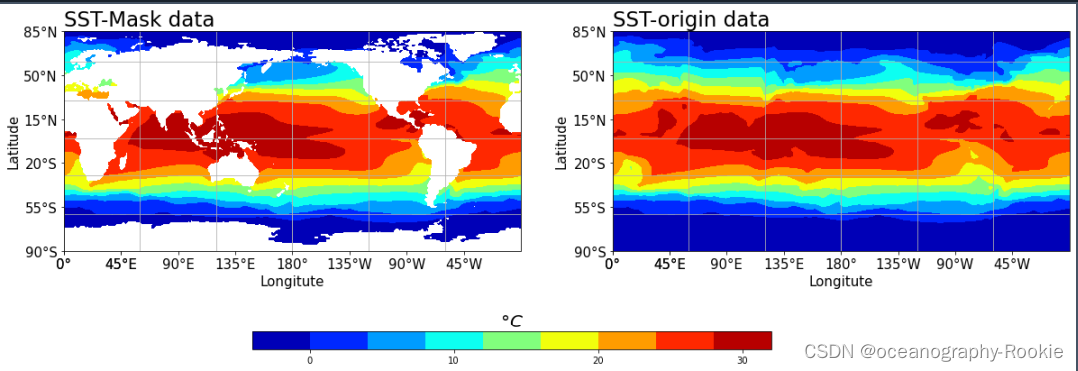
下面以月均海温资料为例,展示掩膜前后的效果:

可以发现,掩膜成功,效果还是不错的。基本满足我们下一步的处理需求。绘图代码如下:
infile = xr.open_dataset(r"sst.mnmean.nc")
infile = mask_land(infile,'land','lon')
infile = infile.transpose("time","lat","lon",...)
def make_map(ax, title):
# set_extent set crs
ax.set_extent(box, crs=ccrs.PlateCarree())
# land = cfeature.NaturalEarthFeature('physical',
# 'land',
# scale,
# edgecolor='face')
ax.set_xticks(np.arange(box[0], box[1] + xstep, xstep),
crs=ccrs.PlateCarree()) # [ )
ax.set_yticks(np.arange(box[2], box[3] + ystep, ystep),
crs=ccrs.PlateCarree())
plt.tick_params(labelsize=15)
lon_formatter = LongitudeFormatter(zero_direction_label=False)
lat_formatter = LatitudeFormatter()
ax.gridlines()
ax.xaxis.set_major_formatter(lon_formatter)
ax.yaxis.set_major_formatter(lat_formatter)
ax.set_ylabel('Latitude', fontsize=15)
ax.set_xlabel('Longitute', fontsize=15)
ax.set_title(title, fontsize=23, loc='left')
return ax
lat = infile['lat'].data
lon = infile['lon'].data
sst = infile['sst'].data
sst_m=np.nanmean(sst,axis=0)
sst_ori = xr.open_dataset(r"sst.mnmean.nc").sst
sst_ori = np.nanmean(sst_ori,axis=0)
box = [0, 361, -90, 85]
scale = '50m'
xstep, ystep = 45, 35
# cmap=plt.get_cmap('rainbow')#'RdYlBu_r'
cmap=cmaps.NCV_jet
fig=plt.figure(figsize=(20,16))
ax=fig.add_subplot(121,projection=ccrs.PlateCarree(central_longitude=180))
make_map(ax,'SST-Mask data')
ax.contourf(lon,lat,sst_m,cmap=cmap,transform=ccrs.PlateCarree(),zorder=2)
ax2=fig.add_subplot(122,projection=ccrs.PlateCarree(central_longitude=180))
make_map(ax2,'SST-origin data')
plot=ax2.contourf(lon,lat,sst_ori,cmap=cmap,transform=ccrs.PlateCarree(),zorder=2)
ax3=fig.add_axes([0.27,0.3,0.4,0.017])
cb=fig.colorbar(plot,cax=ax3,shrink=0.9,ticks=[0,10,20,30],pad=0.04,aspect=15,orientation='horizontal')
cb.ax.tick_params(labelsize=10)
cb.ax.set_title('$°C$', fontsize=20, loc='center')
方法2:使用geopandas+salem库进行掩膜
关于salem和geopandas在Windows系统的安装可以查看以下帖子,可能有用:
使用geopandas+salem进行掩膜相比上述方法,大大减少了代码量,非常方便简洁,只需要提供数据和一个海洋的shp文件即可。缺点在于geopanda和Salem在windows系统的安装较为困难,很难成功安装使用。但是,在linux系统下则相对来说比较容易安装。这里也在Linux系统下以对垂直速度的数据的掩膜进行演示:
我这里的数据的经度排序是0~360,先将其转为-180~180排列,方便我后续处理使用:
import salem
import geopandas as geo
import xarray as xr
ds=xr.open_dataset("omiga.nc")
# change 0-360 to -180 ~ 180
lon_name = 'lon'
ds['longitude_adjusted'] = xr.where(
ds[lon_name] > 180,
ds[lon_name] - 360,
ds[lon_name])
ds = (ds.swap_dims({lon_name: 'longitude_adjusted'}).sel(**{'longitude_adjusted': sorted(ds.longitude_adjusted)}).drop(lon_name))
ds = ds.rename({'longitude_adjusted': lon_name})
# mask
shp_path="shp/10m_physical/ne_10m_ocean_scale_rank.shp"
shp=geo.read_file(shp_path)
sst=ds.salem.roi(shape=shp)
sst.to_netcdf(path="omiga_mask.nc")
绘制填色图查看掩膜结果:
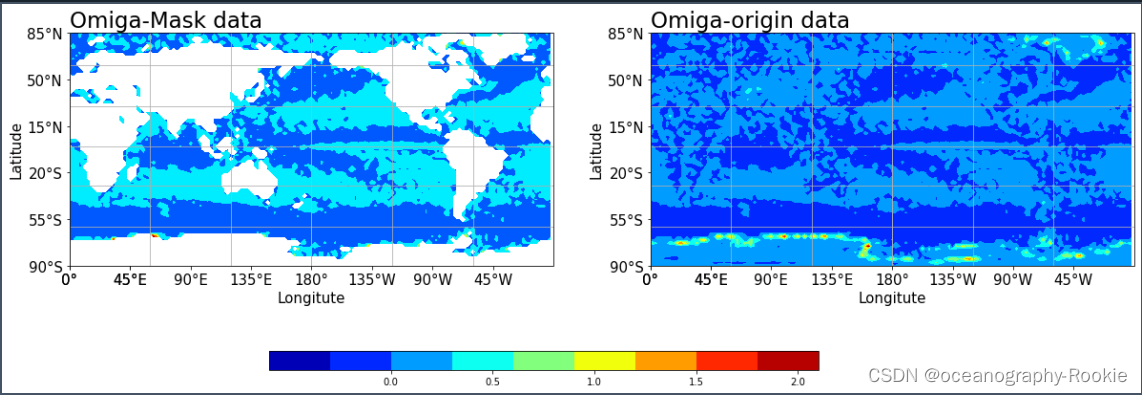
总体上还不错,绘图代码这里就不贴了,与方法1中的代码一致。
虽然不同的掩膜方法掩膜的结果可能不同,但是基本上满足我们后续的计算需求,只需要统一掩膜方法即可。有兴趣的xd可以尝试起来。
一个努力学习python的海洋人
水平有限,欢迎指正!!!
欢迎评论、收藏、点赞、转发、关注。
关注我不后悔,记录学习进步的过程~~
更多推荐
 已为社区贡献16条内容
已为社区贡献16条内容









所有评论(0)