R语言两个矩阵(两组)数据的相关性分析
Mantel tests是确定两组距离测度矩阵(而非两组变量矩阵)之间相关性的相关性测试方法,用于判断一个矩阵中的样本距离与另一矩阵中的样本距离是否相关。Mantel tests零假设为响应变量矩阵中对象之间的距离与解释变量矩阵不存在相关,如果结果中p值显著,则拒绝零假设,即存在相关性,随着一个矩阵中样本之间距离的增加(或减少),另一矩阵中对应样本之间的距离也增加(或减少)。数据是vegan包里自
R语言两个矩阵(两组)数据的相关性分析
vegan包及数据说明
Mantel tests是确定两组距离测度矩阵(而非两组变量矩阵)之间相关性的相关性测试方法,用于判断一个矩阵中的样本距离与另一矩阵中的样本距离是否相关。Mantel tests零假设为响应变量矩阵中对象之间的距离与解释变量矩阵不存在相关,如果结果中p值显著,则拒绝零假设,即存在相关性,随着一个矩阵中样本之间距离的增加(或减少),另一矩阵中对应样本之间的距离也增加(或减少)。
install.packages("vegan")#安装包
library(vegan)#加载包
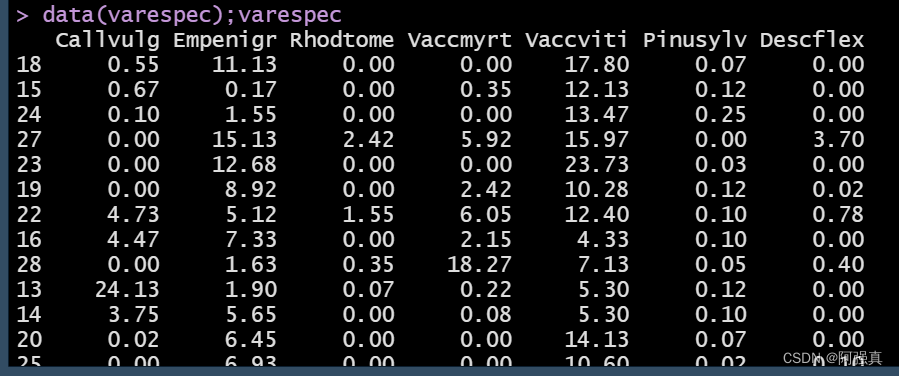
数据是vegan包里自带的两个数据:
data(varespec);varespec
data(varechem);varechem


分别计算距离矩阵
我们使用vegdist函数计算距离矩阵,以下是参数
vegdist(x, method="bray", binary=FALSE, diag=FALSE, upper=FALSE,
na.rm = FALSE, ...)
其中method默认是布雷克蒂斯距离,可以是欧式距离,马氏距离,卡方距离等等
method= “manhattan”, “euclidean”, “canberra”, “clark”, “bray”, “kulczynski”, “jaccard”, “gower”, “altGower”, “morisita”, “horn”, “mountford”, “raup”, “binomial”, “chao”, “cao”, “mahalanobis”, “chisq”, “chord”, “aitchison”, or “robust.aitchison”.
对于这些距离的具体定义可以看这篇文章

veg.dist <- vegdist(varespec))# Bray-Curtis
env.dist <- vegdist(scale(varechem), "euclid")#首先标准化在进行计算
相关性分析
mantel函数进行相关性分析,mantel.partial函数进行偏相关性分析
以下是函数的具体参数
mantel(xdis, ydis, method="pearson", permutations=999, strata = NULL,
na.rm = FALSE, parallel = getOption("mc.cores"))
mantel.partial(xdis, ydis, zdis, method = "pearson", permutations = 999,
strata = NULL, na.rm = FALSE, parallel = getOption("mc.cores"))
具体操作:
mantel(veg.dist, env.dist, method="spear")

这里的significance为0.001小于0.05拒绝原假设H0,即认为两个矩阵之间有很强的相关性
更多推荐
 已为社区贡献7条内容
已为社区贡献7条内容


 华为云 x DeepSeek:AI驱动云上应用创新
华为云 x DeepSeek:AI驱动云上应用创新


 DTT年度收官盛典:华为开发者空间大咖汇,共探云端开发创新
DTT年度收官盛典:华为开发者空间大咖汇,共探云端开发创新


 华为云数字人,助力行业数字化业务创新
华为云数字人,助力行业数字化业务创新


 企业数据治理一站式解决方案及应用实践
企业数据治理一站式解决方案及应用实践


 轻松构建AIoT智能场景应用
轻松构建AIoT智能场景应用








 免费领云主机
免费领云主机



所有评论(0)