详解pandas的read_csv方法
楔子使用pandas做数据处理的第一步就是读取数据,数据源可以来自于各种地方,csv文件便是其中之一。而读取csv文件,pandas也提供了非常强力的支持,参数有四五十个。这些参数中,有的很容易被忽略,但是在实际工作中却用处很大。比如:文件读取时设置某些列为时间类型导入文件, 含有重复列过滤某些列每次迭代指定的行数值替换pandas在读取csv文件是通过read_csv这个函数读取的,下面就来看看
楔子
使用pandas做数据处理的第一步就是读取数据,数据源可以来自于各种地方,csv文件便是其中之一。而读取csv文件,pandas也提供了非常强力的支持,参数有四五十个。这些参数中,有的很容易被忽略,但是在实际工作中却用处很大。比如:
文件读取时设置某些列为时间类型导入文件, 含有重复列过滤某些列每次迭代指定的行数值替换
pandas在读取csv文件是通过read_csv这个函数读取的,下面就来看看这个函数都支持哪些不同的参数,看看它们都生得一副什么模样,是三头六臂,还是烈焰红唇。
read_csv中的参数
下面都是read_csv中的参数,但是根据功能我们划分为不同的类别。
以下代码都在jupyter notebook上运行,Python版本为3.8.2。
基本参数
filepath_or_buffer
数据输入的路径:可以是文件路径、可以是URL,也可以是实现read方法的任意对象。这个参数,就是我们输入的第一个参数。
import pandas as pd pd.read_csv("girl.csv")

还可以是一个URL,如果访问该URL会返回一个文件的话,那么pandas的read_csv函数会自动将该文件进行读取。比如:我们用fastapi写一个服务,将刚才的文件返回。
pd.read_csv("http://localhost/girl.csv")
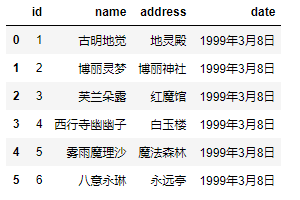
里面还可以是一个 _io.TextIOWrapper,比如:
f = open("girl.csv", encoding="utf-8") pd.read_csv(f)
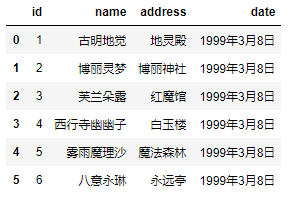
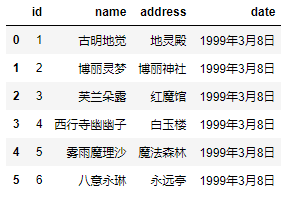
甚至还可以是一个临时文件:
import tempfile import pandas as pd tmp_file = tempfile.TemporaryFile("r+") tmp_file.write(open("girl.csv", encoding="utf-8").read()) tmp_file.seek(0) pd.read_csv(tmp_file)
支持的格式非常齐全,但是一般情况下,我们还是读取实际的csv文件比较多。
sep
读取csv文件时指定的分隔符,默认为逗号。注意:"csv文件的分隔符" 和 "我们读取csv文件时指定的分隔符" 一定要一致。
比如:上面的girl.csv,我们将其分隔符从逗号改成"\t",如果这个时候还是用默认的逗号分隔符,那么数据读取之后便混为一体。
pd.read_csv("girl.csv")

由于指定的分隔符 和 csv文件采用的分隔符 不一致,因此多个列之间没有分开,而是连在一起了。
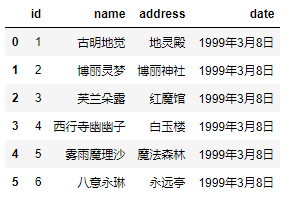
所以,我们需要将分隔符设置成"\t"才可以。
pd.read_csv('girl.csv', sep='\t')
delimiter
分隔符的另一个名字,与 sep 功能相似。
delim_whitespace
0.18 版本后新加参数,默认为 False,设置为 True 时,表示分割符为空白字符,可以是空格、"\t"等等。比如:girl.csv的分隔符是"\t",如果设置delim_whitespace为True的话:
pd.read_csv('girl.csv',delim_whitespace=True)
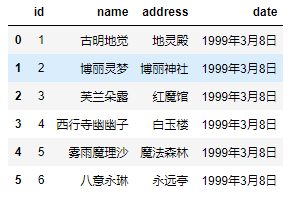
不管分隔符是什么,只要是空白字符,那么可以通过delim_whitespace=True进行读取。
header
设置导入 DataFrame 的列名称,默认为 "infer",注意它与下面介绍的 names 参数的微妙关系。
names
- 当names没被赋值时,header会变成0,即选取数据文件的第一行作为列名。
- 当 names 被赋值,header 没被赋值时,那么header会变成None。如果都赋值,就会实现两个参数的组合功能。
我们举例说明:
- 1) names 没有被赋值,header 也没赋值:
pd.read_csv('girl.csv',delim_whitespace=True) # 我们说这种情况下,header为变成0,即选取文件的第一行作为表头
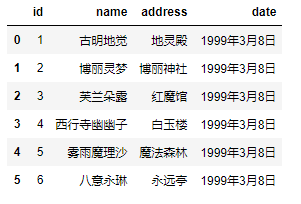
- 2) names 没有被赋值,header 被赋值:
pd.read_csv('girl.csv',delim_whitespace=True, header=1) # 不指定names,指定header为1,则选取第二行当做表头,第二行下面的是数据

- 3) names 被赋值,header 没有被赋值:
pd.read_csv('girl.csv', delim_whitespace=True, names=["编号", "姓名", "地址", "日期"])

我们看到names适用于没有表头的情况,指定names没有指定header,那么header相当于None。一般来说,读取文件会有一个表头的,一般是第一行,但是有的文件只是数据而没有表头,那么这个时候我们就可以通过names手动指定、或者生成表头,而文件里面的数据则全部是内容。所以这里id、name、address、date也当成是一条记录了,本来它是表头的,但是我们指定了names,所以它就变成数据了,表头是我们在names里面指定的
4) names和header都被赋值:
pd.read_csv('girl.csv', delim_whitespace=True, names=["编号", "姓名", "地址", "日期"], header=0)
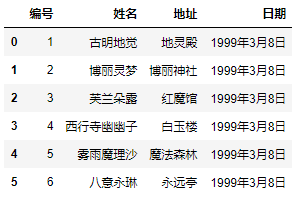
这个相当于先不看names,只看header,我们说header等于0代表什么呢?显然是把第一行当做表头,下面的当成数据,好了,然后再把表头用names给替换掉。
再举个栗子:
pd.read_csv('girl.csv', delim_whitespace=True, names=["编号", "姓名", "地址", "日期"], header=3) # header=3,表示第四行当做表头,第四行下面当成数据 # 然后再把表头用names给替换掉,得到如下结果
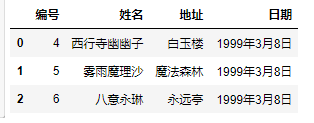
所以names和header的使用场景主要如下:
1. csv文件有表头并且是第一行,那么names和header都无需指定;2. csv文件有表头、但表头不是第一行,可能从下面几行开始才是真正的表头和数据,这个时候指定header即可;3. csv文件没有表头,全部是纯数据,那么我们可以通过names手动生成表头;4. csv文件有表头、但是这个表头你不想用,这个时候同时指定names和header。先用header选出表头和数据,然后再用names将表头替换掉,其实就等价于将数据读取进来之后再对列名进行rename;
index_col
我们在读取文件之后,生成的DataFrame的索引默认是0 1 2 3...,我们当然可以set_index,但是也可以在读取的时候就指定某个列为索引。
pd.read_csv('girl.csv', delim_whitespace=True, index_col="name")
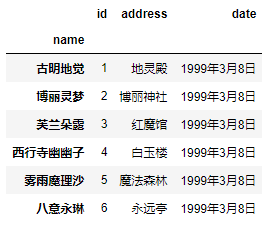
这里指定"name"作为索引,另外除了指定单个列,还可以指定多个列,比如["id", "name"]。并且我们除了可以输入列的名字之外,还可以输入对应的索引。比如:"id"、"name"、"address"、"date"对应的索引就分别是0、1、2、3。
usecols
如果列有很多,而我们不想要全部的列、而是只要指定的列就可以使用这个参数。
pd.read_csv('girl.csv', delim_whitespace=True, usecols=["name", "address"])

mangle_dupe_cols
实际生产用的数据会很复杂,有时导入的数据会含有重名的列。参数 mangle_dupe_cols 默认为 True,重名的列导入后面多一个 .1。如果设置为 False,会抛出不支持的异常:
# ValueError: Setting mangle_dupe_cols=False is not supported yet
prefix
prefix 参数,当导入的数据没有 header 时,设置此参数会自动加一个前缀。比如:
pd.read_csv('girl.csv', delim_whitespace=True, header=None)
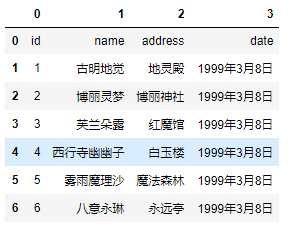
我们看到在不指定names的时候,header默认为0,表示以第一行为表头。但如果不指定names、还显式地将header指定为None,那么会自动生成表头0 1 2 3...,因为DataFrame肯定是要有列名(表头)的。那么prefix参数干什么用的呢?
pd.read_csv('girl.csv', delim_whitespace=True, header=None, prefix="夏色祭")
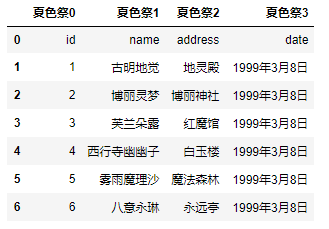
所以prefix就是给这样的列名增加前缀的,个人感觉好像不是很常用,至少本人在工作中从未用过这个参数。
squeeze
感觉又是一个没啥卵用的参数,首先我们读取csv文件得到的是一个DataFrame,如果这个文件只有一列、或者我们只获取一列的话,那么得到的还是一个DataFrame。
pd.read_csv('girl.csv', delim_whitespace=True, usecols=["name"]) # 这里只选择一列

如果指定了squeeze参数为True的话,在只有一列的情况下,那么得到就是一个Series。
pd.read_csv('girl.csv', delim_whitespace=True, usecols=["name"], squeeze=True)

squeeze默认是False,当然如果是多列,即使指定squeeze为True,得到的依旧是DataFrame。如果只有一列,那么本来默认还是DataFrame,但是可以通过指定这个参数为True,将其变成Series。话说你们觉得这个参数有用吗?反正我个人觉得用处不大。
通用解析参数
dtype
笔者就曾遇到一件比较尴尬的事情,就是处理地铁人员数据的。工作人员的id都是以0开头的,比如0100012521,这是一个字符串。但是在读取的时候解析成整型了,结果把开头的0给丢了。这个时候我们就可以通过dtype来指定某个列的类型,就是告诉pandas:你在解析的时候不要自以为是,直接按照老子指定的类型进行解析就可以了,我不要你觉得,我要我觉得。
df = pd.read_csv('girl.csv', delim_whitespace=True) df["id"] = df["id"] * 3 df
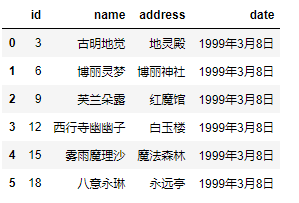
比如这里的id,默认解析的是整型,如果我们希望它是个字符串呢?
df = pd.read_csv('girl.csv', delim_whitespace=True, dtype={"id": str}) df["id"] = df["id"] * 3 df
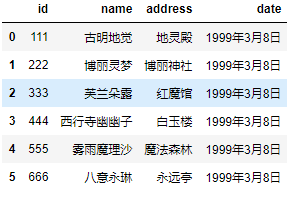
我们看到id变成了字符串类型。
engine
pandas解析数据时用的引擎,pandas 目前的解析引擎提供两种:c、python,默认为 c,因为 c 引擎解析速度更快,但是特性没有 python 引擎全。如果使用 c 引擎没有的特性时,会自动退化为 python 引擎。
比如使用分隔符进行解析,如果指定分隔符不是单个字符、或者"\s+",那么c引擎就无法解析了。我们知道如果分隔符为空白字符的话,那么可以指定delim_whitespace=True,但是也可以指定sep=r"\s+"。
pd.read_csv('girl.csv', sep=r"\s+")
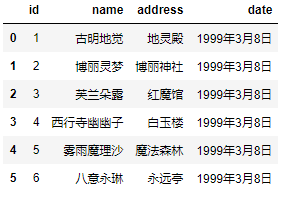
如果sep是单个字符,或者字符串\s+,那么C是可以解决的。但如果我们指定的sep比较复杂,这时候引擎就会退化。
# 我们指定的\s{0}相当于没指定,\s+\s{0}在结果上等同于\s+。 # 但是它不是单个字符,也不是\s+,因此此时的C引擎就无法解决了,而是会退化为python引擎 pd.read_csv('girl.csv', sep=r"\s+\s{0}", encoding="utf-8")

我们看到虽然自动退化,但是弹出了警告,这个时候需要手动的指定engine="python"来避免警告。这里面还用到了encoding参数,这个后面会说,因为引擎一旦退化,在Windows上不指定会读出乱码。这里我们看到sep是可以支持正则的,但是说实话sep这个参数都会设置成单个字符,原因是读取的csv文件的分隔符是单个字符。
converters
可以在读取的时候对列数据进行变换:
pd.read_csv('girl.csv', sep="\t", converters={"id": lambda x: int(x) + 10})
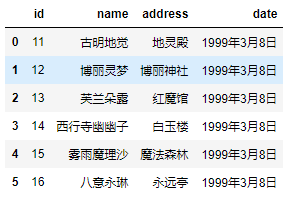
将id增加10,但是注意 int(x),在使用converters参数时,解析器默认所有列的类型为 str,所以需要显式类型转换。
true_values和false_value
指定哪些值应该被清洗为True,哪些值被清洗为False。
pd.read_csv('girl.csv', sep="\t")
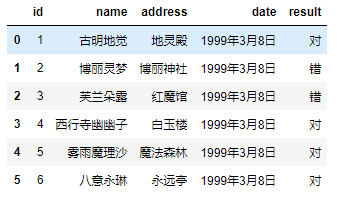
这里增加一个字段result。
pd.read_csv('girl.csv', sep="\t", true_values=["对"], false_values=["错"])
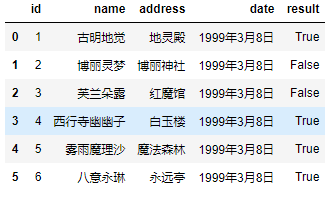
注意这里的替换规则,只有当某一列的数据全部出现在
true_values + false_values里面,才会被替换。
pd.read_csv('girl.csv', sep="\t", false_values=["错"])
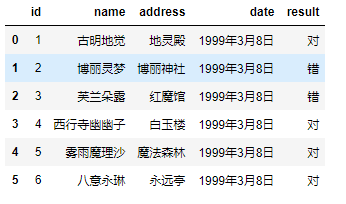
我们看到"错"并没有被替换成False,原因就是只有一个字段中所有的值都在true_values + false_values中,它们才会被替换,而"对"并没有出现。
pd.read_csv('girl.csv', sep="\t", false_values=["错", "对"])

此时就全部被替换成了False。
skiprows
skiprows 表示过滤行,想过滤掉哪些行,就写在一个列表里面传递给skiprows即可。注意的是:这里是先过滤,然后再确定表头,比如:
pd.read_csv('girl.csv', sep="\t", skiprows=[0])

我们把第一行过滤掉了,但是第一行是表头,所以过滤掉之后,第二行就变成表头了。如果过滤掉第二行,那么只相当于少了一行数据,但是表头还是原来的第一行:id、name、address、date、result。
当然里面除了传入具体的数值,来表明要过滤掉哪些行,还可以传入一个函数。
pd.read_csv('girl.csv', sep="\t", skiprows=lambda x: x > 0 and x % 2 == 0)

由于索引从0开始,凡是索引大于0、并且%2等于0的记录都过滤掉。索引大于0,是为了保证表头不被过滤掉。
skipfooter
从文件末尾过滤行,解析引擎退化为 Python。这是因为 C 解析引擎没有这个特性。
pd.read_csv('girl.csv', sep="\t", skipfooter=3, encoding="utf-8", engine="python")

skipfooter接收整型,表示从结尾往上过滤掉指定数量的行,因为引擎退化为python,那么要手动指定engine="python",不然会警告。另外需要指定encoding="utf-8",因为csv存在编码问题,当引擎退化为python的时候,在Windows上读取会乱码。
nrows
nrows 参数设置一次性读入的文件行数,它在读入大文件时很有用,比如 16G 内存的PC无法容纳几百 G 的大文件。
pd.read_csv('girl.csv', sep="\t", nrows=1)
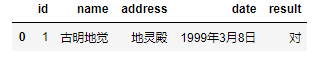
很多时候我们只是想看看大文件内部的字段长什么样子,所以这里通过nrows指定读取的行数。
low_memory
这个看起来是和内存有关的,但更准确的说,其实它是和数据类型相关的。在解释这个原因之前,我们还要先从DataFrame的数据类型说起。
我们知道DataFrame的每一列都是有类型的,那么在读取csv的时候,pandas也是要根据数据来判断每一列的类型的。但pandas主要是靠"猜"的方法,因为在读取csv的时候是分块读取的,每读取一块的时候,会根据数据来判断每一列是什么类型;然后再读取下一块,会再对类型进行一个判断,得到每一列的类型,如果得到的结果和上一个块得到结果不一样,那么就会发出警告,提示有以下的列存在多种数据类型:
DtypeWarning: Columns (1,5,8,......) have mixed types. Specify dtype option on import or set low_memory=False.
而为了保证正常读取,那么会把类型像大的方向兼容,比如第一个块的user_id被解析成整型,但是在解析第二个块发现user_id有的值无法解析成整型,那么类型整体就会变成字符串,于是pandas提示该列存在混合类型。而一旦设置low_memory=False,那么pandas在读取csv的时候就不分块读了,而是直接将文件全部读取到内存里面,这样只需要对整体进行一次判断,就能得到每一列的类型。但是这种方式也有缺陷,一旦csv过大,就会内存溢出。
不过从数据库读取就不用担心了,因为数据库是规定了每一列的类型的。如果是从数据库读取得到的DataFrame,那么每一列的数据类型和数据库表中的类型是一致的。还有,我们在上面介绍了dtype,这个是我们手动规定类型,那么pandas就会按照我们规定的类型去解析指定的列,但是一旦无法解析就会报错。
memory_map
如果你知道python的一个模块mmap,那么你肯定很好理解。如果使用的数据在内存里,那么直接进行映射即可,不会再次进行IO操作,默认为False。这个参数比较底层,我们一般用不到。
空值处理相关参数
na_values
na_values 参数可以配置哪些值需要处理成 NaN,这个是非常常用的,但是用的人不多。
pd.read_csv('girl.csv', sep="\t", na_values=["对", "古明地觉"])

我们看到将"对"和"古明地觉"设置成了NaN,当然我们这里不同的列,里面包含的值都是不相同的。但如果两个列中包含相同的值,而我们只想将其中一个列的值换成NaN该怎么做呢?
pd.read_csv('girl.csv', sep="\t", na_values={"name": ["古明地觉", "博丽灵梦"], "result": ["对"]})
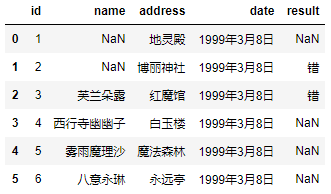
通过字典实现只对指定的列进行替换。
skip_blank_lines
skip_blank_lines 默认为 True,表示过滤掉空行,如为 False 则解析为 NaN
verbose
打印一些额外信息
时间处理相关参数
parse_dates
指定某些列为时间类型,这个参数一般搭配下面的date_parser使用。
date_parser
是用来配合parse_dates参数的,因为有的列虽然是日期,但没办法直接转化,需要我们指定一个解析格式:
from datetime import datetime pd.read_csv('girl.csv', sep="\t", parse_dates=["date"], date_parser=lambda x: datetime.strptime(x, "%Y年%m月%d日"))
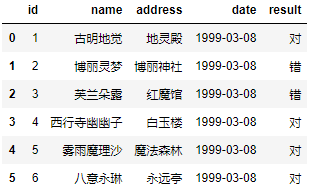
infer_datetime_format
infer_datetime_format 参数默认为 False。如果设定为 True 并且 parse_dates 可用,那么 pandas 将尝试转换为日期类型,如果可以转换,转换方法并解析,在某些情况下会快 5~10 倍。
分块读入相关参数
分块读入内存,尤其单机处理大文件时会很有用。
iterator
iterator 为 bool类型,默认为False。如果为True,那么返回一个 TextFileReader 对象,以便逐块处理文件。这个在文件很大、内存无法容纳所有数据文件时,可以分批读入,依次处理。

chunksize
chunksize 整型,默认为 None,设置文件块的大小。
compression
compression 参数取值为 {'infer', 'gzip', 'bz2', 'zip', 'xz', None},默认 'infer',这个参数直接支持我们使用磁盘上的压缩文件。
# 直接将上面的girl.csv添加到压缩文件,打包成girl.zip pd.read_csv('girl.zip', sep="\t", compression="zip")
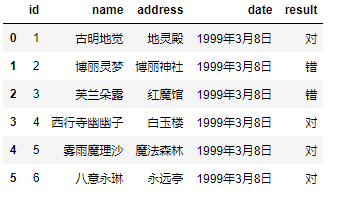
thousands
千分位分割符,如 , 或者 .,默认为None。
encoding
encoding 指定字符集类型,通常指定为 'utf-8'。根据情况也可能是'ISO-8859-1'
error_bad_lines和warn_bad_lines
如果一行包含过多的列,假设csv的数据有5列,但是某一行却有6个数据,显然数据有问题。那么默认情况下不会返回DataFrame,而是会报错。
# ParserError: Error tokenizing data. C error: Expected 5 fields in line 5, saw 6
我们在某一行中多加了一个数据,结果显示错误。因为girl.csv里面有5列,但是有一行却有6个数据,所以报错。
在小样本读取时,这个错误很快就能发现。但是如果样本比较大、并且由于数据集不可能那么干净,会很容易出现这种情况,那么该怎么办呢?而且这种情况下,Excel基本上是打不开这么大的文件的。这个时候我们就可以将error_bad_lines设置为False(默认为True),意思是遇到这种情况,直接把这一行给我扔掉。同时会设置 warn_bad_lines 设置为True,打印剔除的这行。
pd.read_csv('girl.csv', sep="\t", error_bad_lines=False, warn_bad_lines=True)
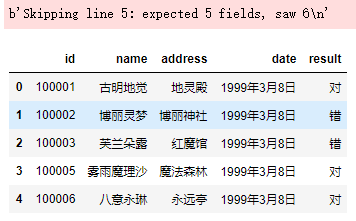
以上两参数只能在C解析引擎下使用。
总结
以上便是pandas的read_csv函数中绝大部分参数了,而且其中的部分参数也适用于读取其它类型的文件。其实在读取csv文件时所使用的参数就那么几个,很多参数平常都不会用,但至少要了解一下,因为在某些特定的场景下它们是可以很方便地帮我们解决一些问题的。
当然,read_csv函数中的参数还不止我们上面说的那些,有几个我们还没有介绍到,感兴趣可以自己看一下。但是个人觉得,掌握上面的那些参数的用法的话,其实已经完全够用了
更多推荐
 已为社区贡献2条内容
已为社区贡献2条内容










所有评论(0)