Can‘t locate Bio/SeqIO.pm in @INC问题极简解决
遇到了Can’t locate Bio/SeqIO.pm in @INC,不能use Bio::SeqIO。尝试了各种方法来解决:一、用如下命令安装bioperl,结果显示不行。$ conda install -c bioconda perl-bioperl二、也采用网上如下步骤用cpan安装bioperl,但是显示有些依赖不能编译成功。强行调用bioperl,不能成功是意料之中。Bioperl的
遇到了Can’t locate Bio/SeqIO.pm in @INC,不能use Bio::SeqIO。极简解决依然是进行正确的@INC环境变量的设置
尝试了各种方法来解决:
一、
用如下命令安装bioperl,没有自动配置好路径,软件依然显示不能运行。
$ conda install -c bioconda perl-bioperl
二、也采用网上如下步骤用cpan安装bioperl,但是显示有些依赖不能编译成功。强行调用bioperl,不能成功。
Bioperl的简单安装
发表于 2013 年 11 月 18 日
按照Bioperl上介绍的方法在linux下安装Bioperl老是安不上,或者是安装上了,但不能用,上面介绍的几种方法都试了,全不行,后面自己想了个办法,就是利用cpan只对要用到的模块进行单独安装,简单适应,如果你和我碰到了同样的问题不妨试试。
0、用root用户登录,不然可能由于权限问题,安装不上去。
1、确定cpan能用。
perl -MCPAN -e shell
cpan>install Bundle::CPAN
2、升级cpan,保证安装的模块是最新的。
cpan>install Module::Build
cpan>o conf prefer_installer MB
cpan>o conf commit
3、安装Bioperl最重要的模块SeqIO(该模块可以实现文件格式转换,计算序列长度,blast信息提取等),中间会有些选项要求选择,一路回车采用默认的就行了。
cpan>install Bio::SeqIO
4、安装SeqFeature模块(序列特征信息的获取或解析)。
cpan>install Bio::SeqFeature
5、安装GenBank模块
cpan>install Bio::GenBank
6、安装AlignIO和AlignI模块(数据格式格式转换)。
cpan>install Bio::AlignIO
cpan>install Bio::AlignI
7、安装DNAstatistics模块(序列统计分析,进化距离计算)。
cpan>install Bio::DNAstatistics
**三、**在服务器中查找SeqIO.pm,发现多处有,都在Bio/文件夹下。cpan和conda都将SeqIO.pm装到了服务器。推测这个Bio/文件夹没有配置到@inc环境变量中。
$ find ./ -name "SeqIO.pm"
$ which perl # 查找perl的路径。
$ perl -V #查perl的@inc的路径配置,没有Bio文件夹。
进行配置@inc环境,重要,不能少!
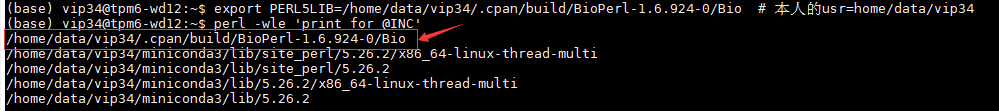
export PERL5LIB=$home/.cpan/build/BioPerl-1.6.924-0/Bio # 本人的$home=home/data/vip34
再perl -wle 'print for @INC'或perl -e 'print join("\n",@INC)',如下图,发现了@inc新环境路径
$ perldoc Bio::SeqIO
出现了如下喜人的信息,死马救活了!

不再是令人失望地出现No documentation found for "Bio::SeqIO",bioperl安装成功!
最后再一劳永逸配置bioperl环境变量,就将如下语句加到~/.bashrc中。以后服务器启动,就自动加载了。
export PERL5LIB=$home/.cpan/build/BioPerl-1.6.924-0/Bio # 本人的$home=home/data/vip34
后面的Bio::SeqFeature、Bio::GenBank、Bio::DNAstatistics和Bio::AlignI 如果调用也出现情况,如法炮制应当即可解决。
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容









所有评论(0)