FastQC安装以及试用
FastQC是一个java程序,能够用于给出测序数据的QC报告,报告中会同时给出上述几个方面的数据图,并提示原来的数据可能还存在着哪些问题,它可以很好地帮助我们理解测序数据的质量情况。使用FastQC需要先有java的环境,在Ubuntu中先使用wget获取安装包(https://download.oracle.com/java/17/latest/jdk-17_linux-x64_bin.tar
·
FastQC是一个java程序,能够用于给出测序数据的QC报告,报告中会同时给出上述几个方面的数据图,并提示原来的数据可能还存在着哪些问题,它可以很好地帮助我们理解测序数据的质量情况。
使用FastQC需要先有java的环境,在Ubuntu中先使用wget获取安装包(https://download.oracle.com/java/17/latest/jdk-17_linux-x64_bin.tar.gz)。
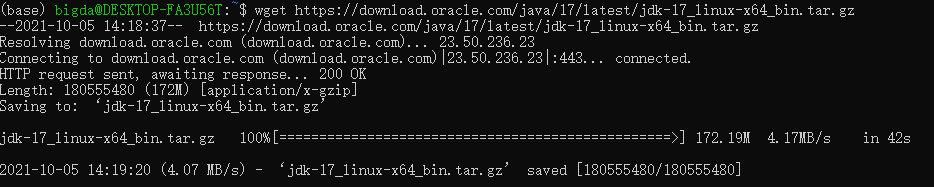
解压之后需要配置环境变量,用自己的安装路径设置JAVA_HOME。
(base) bigda@DESKTOP-FA3U56T:~$ export JAVA_HOME=/home/bigda/jdk-17
(base) bigda@DESKTOP-FA3U56T:~$ export JRE_HOME=$JAVA_HOME/jre
(base) bigda@DESKTOP-FA3U56T:~$ export CLASSPATH=.:$CLASSPATH:$JAVA_HOME/lib:$JRE_HOME/lib
(base) bigda@DESKTOP-FA3U56T:~$ export PATH=$PATH:$JAVA_HOME/bin:$JRE_HOME/bin
完成之后查看一下java的版本,能够查看说明配置完成。

然后去下载FastQC的压缩包(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/fastqc_v0.11.8.zip),使用wget下载完成后解压,再修改文件夹中fastqc的权限,就可以运行了。
(base) bigda@DESKTOP-FA3U56T:~$ unzip fastqc_v0.11.8.zip
(base) bigda@DESKTOP-FA3U56T:~$ cd FastQC
(base) bigda@DESKTOP-FA3U56T:~/FastQC$ chmod 755 fastqc
修改权限就可以看到fastqc从之前的灰色,变成了绿色。


完成后尝试运行,运行的时候,需要给出fastqc文件详细的路径。

等待运行完成后,会在同目录下生成html和zip文件。
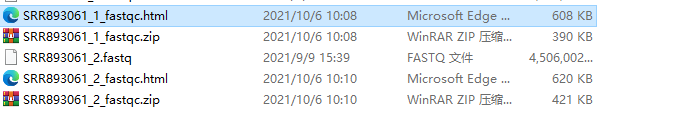
在打开html文件,就可以直观的看到结果。

更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容








所有评论(0)