生信基础 | 人-小鼠同源基因之间的转换
当我们做的转录组或者芯片的数据是小鼠的时候,对于下游的分析,比如GSEA等分析要对生物学数据库注释,发现绝大部分数据库都是人类的基因名字,这个时候我们需要将小鼠的基因映射到人上,就是找小鼠...
·

当我们做的转录组或者芯片的数据是小鼠的时候,对于下游的分析,比如GSEA等分析要对生物学数据库注释,发现绝大部分数据库都是人类的基因名字,这个时候我们需要将小鼠的基因映射到人上,就是找小鼠与人之间的同源基因。有一个取巧的方法是把基因名字修改一下,人的基因名称几乎都是大写,小鼠首字母大写,其他小写。
对于大部分基因来说,直接变换大小写就可以啦。
简单粗暴的方法:
load("K:/BioInfoFiles/hsaGeneInfo.Rdata")#hsaGeneInfo
head(hsaGeneInfo)
library(stringr)
str_to_title(hsaGeneInfo$symbol[1:10])
library(Hmisc)
capitalize(tolower(hsaGeneInfo$symbol[1:10]))

其实有相应的R包(biomaRt)可以进行转换。这里从人到鼠进行转换。
hsa2mus_all <- getLDS(attributes = c("hgnc_symbol"),
filters = "hgnc_symbol",
values = hsaGeneInfo$symbol,
mart = human,
attributesL = c("mgi_symbol"),
martL = mouse,uniqueRows = T)
head(hsa2mus_all)
length(hsaGeneInfo$symbol)
nrow(hsa2mus_all)
> head(hsa2mus_all)
HGNC.symbol MGI.symbol
1 MT-CO1 mt-Co1
2 MT-CO2 mt-Co2
3 MT-ND2 mt-Nd2
4 MT-CO3 mt-Co3
5 MT-ATP8 mt-Atp8
6 MT-ND3 mt-Nd3
> length(hsaGeneInfo$symbol)
[1] 62427
> nrow(hsa2mus_all)
[1] 22616
总共6万多个基因,只有2万多个被映射到小鼠上。这6万多的基因包括了很多假基因。
unique(hsaGeneInfo$gene_type)
hsaGeneInfo$gene_type[match(hsa2mus_all$HGNC.symbol,hsaGeneInfo$symbol)] %>% unique()
只转换一下编码蛋白的基因。
hsa_prot_coding <- hsaGeneInfo$symbol[hsaGeneInfo$gene_type=="protein_coding"]
hsa2mus_protein <- getLDS(attributes = c("hgnc_symbol"),
filters = "hgnc_symbol",
values = hsa_prot_coding ,
mart = human,
attributesL = c("mgi_symbol"),
martL = mouse,uniqueRows = T)
head(hsa2mus_protein)
length(hsa_prot_coding)
nrow(hsa2mus_protein)
发现基因变多了,多出了1000多个。
> length(hsa_prot_coding)
[1] 20068
> nrow(hsa2mus_protein)
[1] 21167
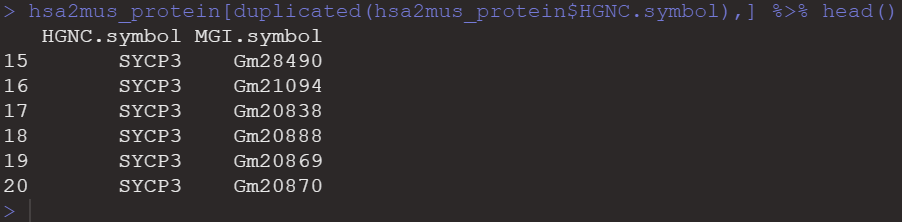
说明一部分人的编码蛋白基因,一个映射到多个小鼠基因上。
hsa2mus_protein[duplicated(hsa2mus_protein$HGNC.symbol),] %>% head()
可以根据前面的得到的对应关系,进行下游的分析。比如GSEA分析。在MSigDB(Molecular Signatures Database)数据库的全部基因集的下载。
http://bioinf.wehi.edu.au/software/MSigDB/human_H_v5p2.rdata
http://bioinf.wehi.edu.au/software/MSigDB/mouse_H_v5p2.rdata
加载这些数据集。将ID进行转换。
library(clusterProfiler)
library(org.Hs.eg.db)
library(org.Mm.eg.db)
load("F:/BioInfoStudy/data/genesets/mouse_H_v5p2.rdata")
load("F:/BioInfoStudy/data/genesets/human_H_v5p2.rdata")
MSigDB_hsa_symb <- lapply(Hs.H, function(x){
bitr(x,
fromType = "ENTREZID",
toType = "SYMBOL",
OrgDb = org.Hs.eg.db)[,2]
})
MSigDB_mus_symb <- lapply(Mm.H, function(x){
bitr(x,
fromType = "ENTREZID",
toType = "SYMBOL",
OrgDb = org.Mm.eg.db)[,2]
})
找出人和小鼠之间的不是简单改变大小写的基因。
h_m_diff <- lapply(names(MSigDB_hsa_symb), function(x){
h = MSigDB_hsa_symb[[x]]
m = toupper(MSigDB_mus_symb[[x]])
hu = setdiff(h,m)})%>% unlist()
通过前面的方法进行转换。
hsa2mus_diff <- getLDS(attributes = c("hgnc_symbol"),
filters = "hgnc_symbol",
values = h_m_diff ,
mart = human,
attributesL = c("mgi_symbol"),
martL = mouse,uniqueRows = T)
head(hsa2mus_diff)
的确,这些基因不是简单的大小写转换:
> head(hsa2mus_diff)
HGNC.symbol MGI.symbol
1 EIF2S3 Eif2s3y
2 H1-6 H1f6
3 H1-2 H1f2
4 SLCO1A2 Gm5724
5 SLCO1A2 Slco1a4
6 SLCO1A2 Slco1a1
参考:
| |
更多推荐
 已为社区贡献3条内容
已为社区贡献3条内容


















所有评论(0)