单细胞测序流程(七)单细胞的细胞类型轨迹分析
系列文章目录单细胞测序流程(一)简介与数据下载单细胞测序流程(二)数据整理单细胞测序流程(三)质控和数据过滤——Seurat包分析,小提琴图和基因离差散点图单细胞测序流程(四)主成分分析——PCA单细胞测序流程(五)t-sne聚类分析和寻找marker基因单细胞测序流程(六)单细胞的细胞类型的注释本期主讲内容——单细胞的细胞类型轨迹分析可以看到细胞的分化从哪里开始,从哪里终止,会分化出什么类型的细
系列文章目录
单细胞测序流程(三)质控和数据过滤——Seurat包分析,小提琴图和基因离差散点图
单细胞测序流程(五)t-sne聚类分析和寻找marker基因
本期主讲内容——单细胞的细胞类型轨迹分析
可以看到细胞的分化从哪里开始,从哪里终止,会分化出什么类型的细胞,或者细胞亚群的分化。
一、课前准备
之前所使用的数据(上个视频中运行结果会有五个txt文件,这就是在所需的数据)
R语言的IDE
二、过程
直接运行代码就可以出现结果,但是我们需要了解绘制这张图的目的和意义
代码如下:
#if (!requireNamespace("BiocManager", quietly = TRUE))
# install.packages("BiocManager")
#BiocManager::install("monocle")
library(monocle)
setwd("在这里写上你文件的地址,不要有中文") #设置工作目录
monocle.matrix=read.table("07.monocleMatrix.txt",sep="\t",header=T,row.names=1,check.names=F)
monocle.sample=read.table("07.monocleSample.txt",sep="\t",header=T,row.names=1,check.names=F)
monocle.geneAnn=read.table("07.monocleGene.txt",sep="\t",header=T,row.names=1,check.names=F)
marker=read.table("07.monocleMarkers.txt",sep="\t",header=T,check.names=F)
#将Seurat结果转换为monocle需要的细胞矩阵,细胞注释表和基因注释表表
data <- as(as.matrix(monocle.matrix), 'sparseMatrix')
pd<-new("AnnotatedDataFrame", data = monocle.sample)
fd<-new("AnnotatedDataFrame", data = monocle.geneAnn)
cds <- newCellDataSet(data, phenoData = pd, featureData = fd)
#给其中一列数据重命名
names(pData(cds))[names(pData(cds))=="seurat_clusters"]="Cluster"
pData(cds)[,"Cluster"]=paste0("cluster",pData(cds)[,"Cluster"])
#添加细胞聚类数据
clusterRt=read.table("07.monocleClusterAnn.txt",header=F,sep="\t",check.names=F)
clusterAnn=as.character(clusterRt[,2])
names(clusterAnn)=paste0("cluster",clusterRt[,1])
pData(cds)$cell_type2 <- plyr::revalue(as.character(pData(cds)$Cluster),clusterAnn)
#伪时间分析流程
cds <- estimateSizeFactors(cds)
cds <- estimateDispersions(cds)
cds <- setOrderingFilter(cds, marker$gene)
plot_ordering_genes(cds)
cds <- reduceDimension(cds, max_components = 2,reduction_method = 'DDRTree')
cds <- orderCells(cds)
pdf(file="cluster.trajectory.pdf",width=6.5,height=6)
plot_cell_trajectory(cds, color_by = "Cluster")
dev.off()
pdf(file="cellType.trajectory.pdf",width=6.5,height=6)
plot_cell_trajectory(cds,color_by = "cell_type2")
dev.off()三、结果

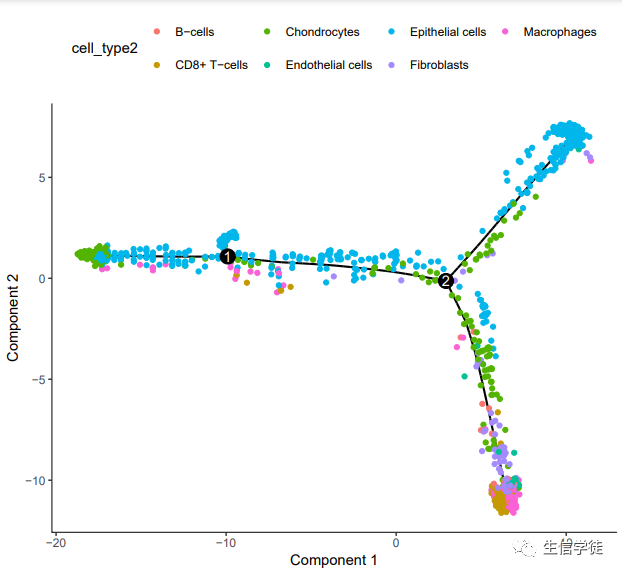
这张图的上方我们可以看到每个细胞所代表的颜色,再从图中看结果,从1前面的地方开始,绿色所代表的细胞是最先开始的,这代表细胞最初的类型,经过细胞分化逐渐有其他的细胞,到2的时候开始分类,向下会分化成更多类型的细胞,向上就基本不变。举个例子:在研究癌症细胞分化时,我们不知道这个细胞来源于哪里也不知道他会分化成什么,但是可以根据这张图就可以知道大致会分化为什么细胞和大概来源于什么细胞。
这与上图不同的是,这张图是各个细胞亚群的轨迹分析,它可以看出每个细胞亚群的分化,但是轨迹图基本与上图相同。我们可以根据每一个细胞亚群的细胞类型与上图一一对照。
结尾
因为这次的结果很多取决于之前的数据,所以必须要把上一节课的内容也要用到,所以要保证之前所得到结果无误才可以。
单细胞测序流程(七)单细胞的细胞类型的细胞轨迹分析到这里就已结束了
下一章会讲解maeker基因转化和富集分析,这次很多取得的数据都会用于下次课程不要删除哦。
我所做的所有分析与教程的代码都会在我的个人公众号中,请打开微信搜索“生信学徒”进行关注,欢迎生信的研究人员和同学前来讨论分析。
ps:公众号刚刚建立比较简陋,但是该有的内容都不会少。
更多推荐
 已为社区贡献6条内容
已为社区贡献6条内容









所有评论(0)